これは、ケルシー・フロレク博士とカーティス・カプサック博士からのゲストブログ投稿です。 からのこの投稿のビデオプレゼンテーション Dockerのコミュニティオールハンズ 見つけることができます ここは.
ケルシー・フロレク博士は、ウィスコンシン州立衛生研究所のシニアゲノミクスおよびデータサイエンティストであり、州公衆衛生バイオインフォマティクスグループ(StaPH-B)の運営委員会メンバーです。
Curtis Kapsakは、Theiagen Genomicsのバイオインフォマティクス科学者であり、州公衆衛生バイオインフォマティクスグループ(StaPH-B)のメンバーです。
2019年の新しい重症急性呼吸器症候群(SARS)ウイルスの急速な出現と世界的な蔓延により、公衆衛生研究所はこれまでにない規模でゲノムモニタリングの取り組みのための新しい方法を開発するようになりました。 この課題に加えて、ゲノムデータ解析で一般的に使用されるアプローチは、分析パイプラインやワークフローの設定の複雑さを増す最先端の、そしてしばしばニッチなオープンソースソフトウェアとライブラリに依存することがよくあります。 これは、オンプレミスのワークステーションからパブリッククラウドに至るまでのコンピューティング環境のさまざまな状況とともに、ウイルスゲノムモニタリングを実行しようとする多くのラボにとって大きな障壁を生み出しました。
ドッキングされたツールのライブラリの開発
公衆衛生研究所は、本質的に厳格な品質管理と品質保証基準を満たす必要があります。 公衆衛生研究所で実施された検査は、患者のケアに使用されるために診療所に報告されるか、公衆衛生介入または発生調査に情報を提供するためにまとめて使用されます。 分析ワークフローは、他のラボで開発されたテストと同じ基準に保たれており、この取り組みをサポートするために、 州公衆衛生バイオインフォマティクス コンソーシアム(StaPH-B)は、公衆衛生ゲノムデータ分析で一般的に使用されるドッキングされたソフトウェアのリポジトリである StaPH-B / docker-buildsの開発を開始しました。 このリポジトリは、信頼性と再現性の両方を備えたアクセス可能なソフトウェアのニーズに対応するように設計されています。 使用ガイドと組み合わせることで、このリポジトリは、分析ワークフローを開発するラボをサポートするために、保守およびテストされたオープンソースツールの一元化された場所を提供しました。
2018年の最初の開発以来、 StaPH-B / docker-build リポジトリは、19の異なる貢献者からの90を超える異なる分析ツールの複数のバージョンを含むように成長し、COVID-19固有の画像のいくつかは100万を超えるプルを達成しました。 2021年3月から2022年1月の間に、より多くのラボがゲノムモニタリングを開始するにつれて、コアCOVID-19ゲノム解析ソフトウェアのDocker画像プルの数が対数的に増加しました。
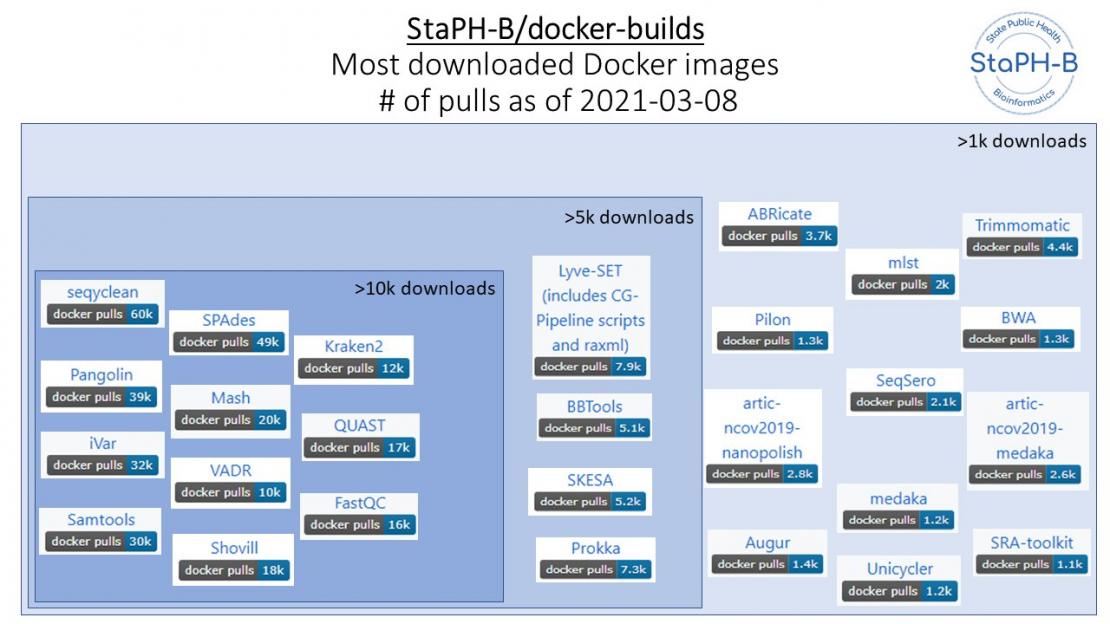

COVID-19ゲノム解析のサポート
バイオインフォマティクスのパイプラインやワークフローは、さまざまなツールで構成されており、多くの場合、生または一次DNAシーケンシングデータの形式から始まります。 これらのツールは、さまざまな変換タスクまたはサマリータスクを実行し、計算要件と依存関係の両方が異なります。 SARS-CoV-2ウイルスゲノムの配列決定プロセスには、ウイルスゲノムを切断し、DNAのごく一部を並行して配列決定することが含まれます。 その結果、さまざまなシーケンスの組み合わせで A、T、C、G を含む数十万から数百万の短い文字列を含むデータセットが作成されます。 次に、COVID-19ワークフローはこれらのデータセットを取得し、ゲノムを再構築し、さまざまな手法を使用してウイルスを特徴付けます。
世界中の多くのラボは、分析ワークフローに WDL や Nextflow などの専用のワークフロー言語を使用する方向に移行しています。 ワークフロー言語と Docker 化されたソフトウェアを組み合わせることで、移植性が高く、さまざまなコンピューティング環境に簡単に適応できるワークフローの作成と日常的な使用が可能になります。 これにより、ラボはラップトップで小規模なデータセットを実行したり、大規模なデータセット用に高性能コンピューティングクラスターまたはクラウド環境に拡張したりできます。 さらに、これらのワークフローアプローチにより、新しいバージョンがリリースされたり、問題が特定されたりしたときにソフトウェアをスワップアウトできるモジュラー分析フレームワークを開発できます。 COVID-19を引き起こすウイルスの急速かつ絶え間ない進化に伴い、分類ソフトウェアの更新も頻繁に更新され、バリアントを正確に識別する能力を維持しています。
ウイルスの進化に遅れずについていく
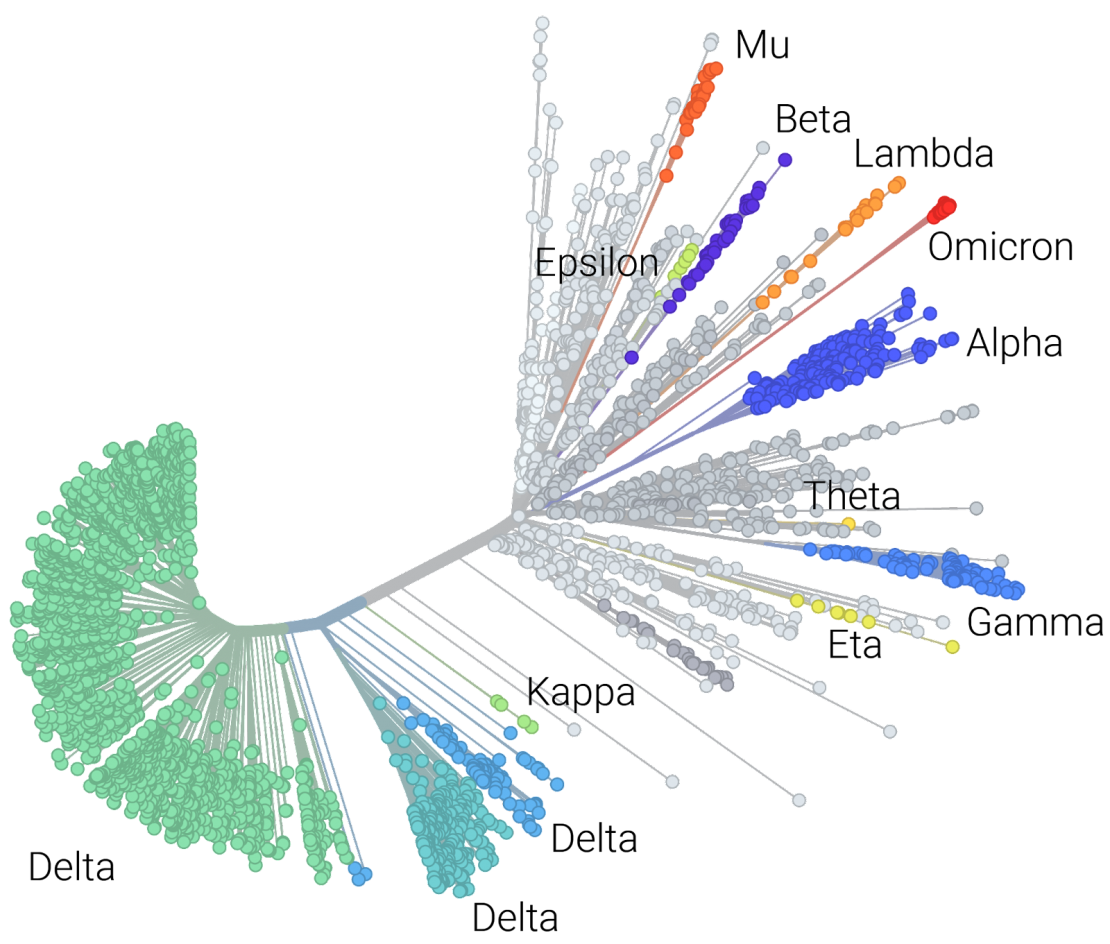
COVID-19ウイルスは、月に平均2つの突然変異が発生するインフルエンザよりも少し遅く進化し、さまざまな変異体(アルファ、デルタ、オミクロンなど)が突然変異のさまざまな組み合わせによって区別されます。 ウイルスを分類するには、新しいウイルスと他のウイルスとの関係をモデル化する系統樹を構築する必要があります。 ただし、新しいウイルスを以前のすべてのウイルスと比較するツリーを構築することは、計算コストが高く、実用的ではありません。 これに対処するために、選択された参照ウイルスのセットを使用してツリーを構築する(Nextclade)か、機械学習を使用して突然変異パターンを分類する(センザンコウ)など、一般的に使用される2つの方法が登場しました。 どちらのアプローチも、最新の情報で分類が正確に行われていることを確認するために、定期的な更新が必要です。 コンテナ化を活用することで、 StaPH-B は最新のモデルで画像を維持することができ、ユーザーは最新の堅牢でテスト済みの分類ツールを使用していることを知ってワークフローを実行できます。
概要
コンテナリゼーションの移植性が高く、スケーラブルで効率的な性質により、公衆衛生上の病気のモニタリングの実行方法が変わりました。 コンテナ化されたワークフローの実装により、ラボは複雑な分析ワークフローを迅速に採用できるようになり、ウイルスモニタリングの取り組みの規模が拡大しました。 StaPH-Bが維持するオープンソースリポジトリは、イノベーションを推進するバイオインフォマティシャンのコミュニティなしでは不可能です。 シーケンシングや複雑な分析に目を向けるラボが増えるにつれ、生物学と情報学の間のギャップを埋める人々に対する需要が高まっています。 バイオインフォマティクスのキャリアとデータを使用して健康問題を解決することに興味がある場合は、APHL-CDCバイオインフォマティクスフェローシップをチェックしてください。
ドッカーコン2022
5月10日火曜日に開催されるDockerCon2022にご参加ください。 DockerCon は、次世代の最新アプリケーションを構築している開発者や開発チームにとってユニークな体験を提供する、無料の 1 日の仮想イベントです。 コードからクラウドにすばやく移行する方法と開発の課題を解決する方法について学びたい場合は、DockerCon 2022 でアプリケーションの構築、共有、実行に役立つ魅力的なライブ コンテンツが提供されます。 今すぐご登録ください https://www.docker.com/dockercon/
